2.辽宁省农业科学院, 沈阳, 110161
3.中国农科院蔬菜花卉研究所, 北京, 100081
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 24 篇 doi: 10.5376/mpb.cn.2011.09.0024
收稿日期: 2011年01月11日 接受日期: 2011年02月28日 发表日期: 2011年03月02日
陈丽静等, 2011, 百合SRAP-PCR反应体系的建立与优化, 分子植物育种 Vol.9 No.24 (doi: 10.5376/mpb.cn.2011.09.0024)
建立适宜百合DNA的SRAP-PCR扩增体系,为百合基因图谱的构建和分子标记打下基础。以百合为试验材料,试验在确定了模板浓度和引物浓度后,采用二因素交叉试验方法,对影响百合SRAP反应体系的5种因素(模板DNA、引物、Mg2+浓度、dNTPs、Taq聚合酶用量)进行优化筛选,建立了适合百合的扩增多态性高、重复性好、带型清晰的最佳SRAP-PCR反应体系,即在15μL PCR反应体系中含有1.5 μL 10×PCR Buffer、80 ng模板DNA、0.11 μmol﹒L-1引物、1.67 mmol﹒L-1 Mg2+、0.267 mmol﹒L-1 dNTPs、1.0 U Taq聚合酶,PCR产物变性时用10 μL变性剂。
SRAP (Sequence-related amplified polymorphism,相关序列扩增多态性)分子标记技术是由美国加州大学Li和Quiros博士(2001)在2001首先提出,也称为基于序列扩增多态性(Sequence-based amplified polymorphism,SBAP)(Li et al., 2003),具有简单、高效、高共显性、重复性、易测序等优点,适合应用于遗传图谱构建、基因定位与克隆、比较基因组学、遗传多样性分析等研究领域(Ferriol et al., 2003; Li et al., 2003; Li et al., 2001)。目前已经在辣椒、油菜、大蒜、莴苣、芹菜、马铃薯、苹果、柑橘、樱桃、梅子、棉花、小麦和水稻等植物研究中应用(Ferriol et al., 2003; Li et al., 2003; Li et al., 2001;李丽等, 2006; 李媛媛等, 2007; 林忠旭等, 2003; 林忠旭等, 2003; 任羽等, 2004; 吴伟怀等, 2004; 武志朴等, 2005)。
SRAP分子标记是基于PCR的标记系统,因此PCR中模板DNA、引物、Mg2+浓度、dNTPs、Taq聚合酶用量以及PCR中变性、退火、延伸的温度和时间等因素均会对结果产生影响。本研究拟通过建立百合SRAP-PCR反应体系并对其进行优化,以探索新型分子标记技术SRAP在百合遗传图谱构建及分子标记辅助育种研究中应用的可行性,为百合优质种质资源的科学利用、分子辅助育种和品种鉴定奠定技术基础。
1 结果与分析
1.1 模板浓度对PCR结果的影响
不同模板DNA浓度对SRAP 扩增体系有明显影响,由图1可以看出当模板DNA的量低于20 ng时,扩增产物的谱带相对较少,且谱带较淡,这是因为模板的量太少,模板与引物不能有效配对,扩增效率低;而大于40 ng时,SRAP产物基本保持不变,说明SRAP扩增对模板DNA的浓度范围可以较宽。
本试验模板DNA用量在10 ng到90 ng之间均能够扩出较为清晰的条带,但模板量在高于80 ng和低于40 ng时分别出现高分子量条带的模糊和中分子量条带的缺失。推测模板量太低,影响扩增产物的形成,而过多的模板DNA在扩增产物积累到一定量时会限制反应。通过比较,本研究选用80 ng为最佳模板。
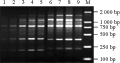 图1 DNA 浓度对SRAP 反应的影响 注: M: DL2000 DNA maker; 1: 10 ng DNA/15 μL; 2: 20 ng DNA/15 μL; 3: 30 ng DNA/15 μL; 4: 40 ng DNA/15 μL; 5: 50 ng DNA/15 μL; 6: 60 ng DNA/15 μL; 7: 70 ng DNA/15 μL; 8: 80 ng DNA/15 μL; 9: 90 ng DNA/15 μL Figure 1 The effects of DNA concentration on SRAP patterns Note: M: DL2000 DNA maker; 1: 10 ng DNA/15 μL; 2: 20 ng DNA/15 μL; 3: 30 ng DNA/15 μL; 4: 40 ng DNA/15 μL; 5: 50 ng DNA/15 μL; 6: 60 ng DNA/15 μL; 7: 70 ng DNA/15 μL; 8: 80 ng DNA/15 μL; 9: 90 ng DNA/15 μL |
1.2 引物浓度梯度试验结果
结果见图2。从反应结果来看最佳引物浓度十分重要:较低的引物浓度(A1、A2、B1、B2所示条带),引物与模板的结合概率下降,扩增条带浅,有的甚至检测不出来。较高的引物浓度(A8、A9、B8、B9号带)扩增的条带整齐清晰,效果较好。适中的引物浓度(A3、A4、B3、B4号带)扩增的条带整齐清晰,效果也较好,考虑到较高的引物浓度会引起引物二聚体的产生,确定引物浓度为0.11 μmol•L-1。
|
图2 引物浓度对SRAP反应的影响 注: 引物浓度设置见表2; M: DL2000 DNA maker Figure 2 The effects of primer concentrations on SRAP Note: primer concentrations designed in table2; M: DL2000 DNA marker |
1.3 最佳Mg2+浓度的确立
结果如图3所示。在引物浓度相同时,随着Mg2+浓度的增加,产物量先增加后减少。考虑到Mg2+浓度过低不利于酶的激活,Mg2+浓度太高又容易产生非特异性扩增,故试验最终选定Mg2+浓度为1.67 mmol•L-1。
 图3 Mg2+和引物浓度对SRAP反应的影响 注: Mg2+和引物浓度设置见表2; M: DL2000 DNA maker Figure 3 The effects of Mg2++concentrations on SRAP Note: Mg2+ and primer concentrations designed in table4; M: DL2000 DNA marker |
1.4 dNTP浓度对PCR结果的影响
Mg2+浓度为1.67 mmol•L-1的情况下,随着dNTPs浓度的增加,PCR扩增产物的量逐渐增加;同时非特异性带也随之增加;当Mg2+浓度为0.83mmol•L-1时,随着dNTP浓度的增加,扩增产物先增加后减少。这与前人的研究不完全一致(李严等, 2005)。本试验最终选择dNTP浓度为0.267 mmol•L-1,Mg2+浓度为1.67mmol•L-1。结果如图4所示。
.png) 图4 dNTPs浓度对SRAP反应的影响 注: 引物浓度设置见表2, M: DL2000 DNA maker Figure 4 The effects of dNTPs concentrations on SRAP Note: dNTPs concentrations designed in table5; M: DL2000 DNA marker |
1.5 Taq 聚合酶的用量对PCR结果的影响
试验表明,Taq酶量在0.5 U-2 U/15 μL反应体系时都能得到可重复的清晰条带,基于经济原因考虑本试验最终选择采用1 U/15 μL反应体系,结果见图5。
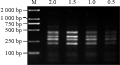 图5 Taq DNA聚合酶的纯度和用量对SRAP反应的影响 注: 酶浓度从左至右依次为2.0, 1.5, 1.0, 0.5; M: DL2000 DNA maker Figure 5 The effects of Taq DNA Polymerase Note: Taq DNA Polymerase concentrations 2.0, 1.5, 1.0, 0.5; M: DL2000 DNA marker |
1.6退火温度和循环次数对扩增的影响
退火温度的高低影响PCR的特异性,退火温度越高,扩增的特异性越高,本试验按照Li等(2001)的反应程序对百合DNA进行扩增,电泳结果显示扩增条带不够清晰。因此设计了退火温度梯度试验:采用了在一个循环体系中采用一低一高两个不同退火温度,首先在34℃的退火温度下扩增5次循环,然后适当提高退火温度在50℃扩增35个循环。可以得到比较理想的扩增结果如图6所示。
|
图6 循环程序对SRAP 反应的影响 注: 1-6采用循环程序1; 7-12采用循环程序2; M: DL2000 DNA maker Figure 6 The effects of circles program on SRAP patterns Note: 1-6 adopted circles program1; 7-12 adopted circles program2; M: DL2000 DNA marker |
优化后的程序为: 94℃ 5 min, 94℃ 1 min,34℃ 1 min,72℃ 1 min, 5 cycles;接下来将复性温度提高到50℃,94℃ 1 min,50℃ 1 min,72℃ 1 min,35 cycles,最后72℃ 5 min,4℃保存。从图6中可以看出能够扩增出清晰稳定的条带,并有较好的多态性。
1.7 SRAP-PCR 体系及反应参数的稳定性检测结果
选择SRAP引物组合me9+em8进行PCR,电泳结果如图7所示,所选择的引物能扩增出清晰、重复性好的条带,表明优化的反应体系参数稳定可靠。
2讨论
研究表明,不同种的试验材料PCR扩增反应体系和扩增程序不同,大多数研究者进行SRAP研究时都会都会对PCR扩增反应体系与扩增程序进行优化,但是通常他们的优化方法都是采用正交实验的方法(杨琦等, 2007; 王振国, 2007; 郭大龙, 2008)。正交实验的优点为:完成试验要求所需的实验次数少,数据点的分布很均匀,可用相应的极差分析方法、方差分析方法、回归分析方法等对试验结果进行分析,引出许多有价值的结论。但是其缺点也很明显,即有可能得到的实验条件并不是最优条件。所以本试验在确定了模板浓度和引物浓度后,主要采用二因素方法确定剩下的反应条件。虽然会加大一定的工作量,但得到的结果也更加准确可靠。
SRAP是一种不需要内切酶消化的分子标记技术,通常适宜植物PCR的模板DNA量为1-100 ng。SRAP的产生是由于遗传材料的基因组DNA在特定引物结合区域产生DNA片段的插入、缺失或碱基突变后可能导致PCR产物增加、缺少或发生变化,产生特异的条带。因此微量的DNA差异也可能导致PCR产物的变化。汪小全等(1996)认为模板浓度在一个相当大的范围内不影响扩增结果,而模板降解程度则会对SRAP的结果产生影响。本试验支持汪小全等的观点。
目前在SRAP-PCR中使用2种扩增程序。第1种由Li等(2001)提出的即最初5个循环的退火温度设为35℃。后30个循环的退火温度升高为50℃。第2种是Budak等(2004)提出的:94℃变性1 min,47℃退火1 min,72℃延伸1 min (35个循环),72℃ 5 min。本试验采用了在一个循环体系采用一低一高两个不同退火温度,扩增效果较为理想。
关于SRAP的反应体系和程序优化的报道(任羽等, 2004; 武志朴等, 2005)很多,其中模板DNA、Mg2+浓度、引物浓度、dNTPs、Taq DNA聚合酶等各组分的用量会影响扩增结果。本试验在优化SRAP扩增体系时发现,Mg2+、引物、dNTPs、Taq DNA聚合酶作为SRAP扩增的底物,有一个相对适宜的用量范围,浓度过低,不能满足扩增要求;浓度过高,组分间可能会产生竞争,降低各组分的活性,影响扩增效果。因此,构建同一物种在相同仪器设备和一定操作规范下的最佳的优化技术体系是进行SRAP研究的关键所在。
3 材料与方法
3.1 试验材料
亲本材料:采用实验室胚培养的“雷山一号”与“晶体”做为父母本材料低温冰箱保存备用。
群体材料:采集180株RIL群体新生幼嫩叶片低温冰箱保存备用。
3.2 基因组DNA的提取
参照黄永芳等(2006)提出的改良的CTAB法提取百合群体基因组DNA。用紫外分光光度计测定DNA样品的质量和浓度,并于1%琼脂糖凝胶检测。最后将纯化后的DNA稀释到200 ng/μL用于后续试验。
3.3 PCR反应
PCR反应在BIO-RAD公司的PTC-100 PCR仪中进行,扩增程序为:94℃ 预变性5 min,94℃ 1 min,34℃ 1 min,72℃ 1 min,5 cycles,94℃ 1 min,50℃ 1 min,72℃ 1 min,35 cycles,最后72℃ 5 min,4℃保存。PCR反应总体积是15 μL。PCR产物采用6%变性聚丙烯酰胺凝胶电泳,Na2CO3银染法染色显影,在荧光灯上观察分析条带。
3.4 PCR反应体系优化的二因素交叉试验设计
3.4.1 模板浓度对PCR结果的影响
不同模板DNA浓度对SRAP 扩增体系有明显影响,模板浓度梯度设置如表1(进行两次重复)所示。
3.4.2 引物浓度梯度试验
最佳引物浓度十分重要,在确定模板浓度之后,引物浓度其浓度梯度设置如表2(进行两次重复)所示。为了保证电泳和检测的的效果,采用的是变性聚丙烯酰胺凝胶电泳并用银染法进行检测。SRAP所用引物序列如表3所示。
3.4.3 最佳Mg2+浓度的确立
最佳引物浓度与最佳Mg2+浓度的确立显得十分重要,考虑到引物浓度与Mg2+浓度互作的影响,在初步确定引物浓度之后,在前期所做的工作基础上,开展引物浓度和Mg2+浓度的二因素交叉实验,引物组合采用me10+em4,浓度梯度设计如表4(重复两次, A, a重复, B,b重复, C, c重复)所示。
3.4.4 dNTP浓度对PCR扩增的影响
针对dNTP浓度和Mg2+的互作效应,进行了二因素试验设计,浓度梯度设置见表5。
反应体系:
Taq Enzyme 0.3 μL
Taq Buffer 1.5 μL
Mg2+ 1.8 μL
dNTP Mix 1.2 μL
PrimersFR 1.5+1.5 μL
DNA 1.5 μL
ddH2O 5.7 μL
3.5优化体系的稳定性检测
选择另外5对SRAP引物作为检测引物,用优化的百合SRAP反应体系,对百合基因组DNA进行PCR扩增,每对引物重复3次。扩增产物经变性聚丙烯酰胺凝胶电泳后观察结果。检测引物的序列如表6。
4结论
本试验利用聚丙烯酰胺凝胶电泳的方法研究百合SRAP标记的最佳反应体系和反应程序。试验结果表明,百合SRAP 分析的最优反应体系为:在15μL反应体系中模板为80 ng,引物浓度为0.11μmol•L-1,Mg2+浓度为1.67 mmol•L-1,dNTP 浓度为0.267 m mol•L-1,1 U的Taq酶。百合SRAP 分析的最佳反应程序为:94℃ 1 min,34℃ 1 min,72℃ 1 min,5个循环,94℃ 1 min,50℃ 1 min,72℃ 1 min,35个循环。
作者贡献
作者陈丽静负责本文初稿的执笔,曹珊负责本文初稿的编辑及反复修改,王玉坤和刘烜晨做的聚丙烯酰胺凝胶电泳,张丽引物的筛选,明军提供实验材料及文章的修改思路,陶承光和李天来提供整体科研的思路,经费和文章的修改。
致谢
本研究由辽宁省教育厅项目(L2010495)、中国博士后科学基金面上项目(20100471471)、国家高技术研究发展计划(863计划)项目(2006AA100109)资助。作者感谢天根公司刘玉东在本实验过程中的技术支持和有益的建议。感谢同行评审人的评审建议和修改建议。
参考文献
Budak H., Shearman R.C., and Parmaksiz I., 2004, Molecular characterization of Buffalograss germplasm using sequence-related amplified polymorphism markers, Theor. Appl. Genet, 108: 328-324 doi:10.1007/s00122-003-1428-4 PMid:13679978
Ferriol M., Pico B., and Nuez F., 2003, Genetic diversity of some accessions of Cucurbita maxima from Spain using RAPD and SRAP markers, Genetic Resources and Crop Evolution, 50(3): 227-238 doi:10.1023/A:1023502925766
Guo D.L., Hou X.G., Zhang J., and Han L., 2008, The orthogonal optimization of SRAP amplification system in Paeonia suffruticosa, Henan Nongye Kexue (Scientia Agricultura Henan), 12: 110-113 (郭大龙, 侯小改, 张静, 韩璐, 2008, 牡丹SRAP反应体系的建立及正交设计优化, 河南农业科学, 12: 110-113)
Huang Y.F., Yang M.X., Liu J., and Zhou J.P., 2006, DNA extraction and optimization of RAPD reaction system for Lilium brownii, Redai Yaredai Zhiwu Xuebao (Journal of Tropical and Subtropical Botany), 14(3): 251-255 (黄永芳, 杨懋勋, 柳军, 周锦平, 2006, 广东野百合DNA提取和RAPD条件的优化, 热带亚热带植物学报, 14(3): 251-255)
Li G., and Quiros C.F., 2001, Sequence-related amplified polymorphism (SRAP),a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica, Theor. Appl. Genet., 103:455-461 doi:10.1007/s001220100570
Li G., Gao M., and Yang B., 2003, Gene for gene alignment between the Brassica and Arabidopsis genomes by direct transcriptome mapping, Theor. Appl. Genet., 107: 271-282
Li L., Zheng X.Y., and Liu L.W., 2006, Analysis of genetic diversity and identification of Cucumber varieties by SRAP, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 4(5): 702-708 (李丽, 郑晓鹰, 柳李旺, 2006, 用SRAP标记分析黄瓜品种遗传多样性及鉴定品种, 分子植物育种, 4(5): 702-708)
Li Y., and Zhang C.Q., 2005, A molecular marker-SRAP technique optimization and application analysis, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 21(5): 108-112 (李严, 张春庆, 2005, 新型分子标记-SRAP技术体系优化及应用前景分析, 中国农学通报, 21(5): 108-112)
Li Y.Y., Shen J.X., wang t.h., Fu T.D., and Ma C.Z., 2007, Construction of a linkage map using SRAP, SSR and AFLP markers in Brassica napus L., Zhongguo Nongye Kexue (Scientia Agricultura Sinica), 40(6): 111-1126 (李媛嫒, 沈金雄, 王同华, 傅廷栋, 马朝芝, 2007, 利用SRAP、SSR和AFLP标记构建甘蓝型油菜遗传连锁图谱, 中国农业科学, 40(6): 1118-1126)
Lin Z.X., Zhang X.L., and Nie Y.C., 2003, Construction of Cotton SSR-AFLP based genetic linkage map, Kexue Tongbao (Chinese Sci. Bull.), 48(5): 1676-1679 (林忠旭, 张献龙, 聂以春, 2003, 棉花SRAP遗传连锁图构建, 科学通报, 48(5): 1676-1679)
Lin Z.X., Zhang X.L., and Nie Y.C., 2004, Evaluation of application of a new molecular marker SRAP on analysis of f2 segregation population and genetic diversity in Cotton, Yichuan Xuebao (Acta Genetica Sinica), 31(6): 622-626 (林忠旭, 张献龙, 聂以春, 2004, 新型标记SRAP在棉花F2分离群体遗传多样性评价中的适用性分析, 遗传学报, 31(6): 622-626)
Ren Y., Wang D.Y., and Zhang Y.D., 2004, Sequence-related amplified polymorphism(SRAP): A novel technique for molecular marker, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 209(6): 11-13, 22 (任羽, 王得元, 张银东, 2004, 相关序列扩增多态性(SRAP)一种新的分子标记技术, 中国农学通报, 209(6): 11-13, 22)
Ren Y., Wang D.Y., Zhang Y.D., Li Y., and Wang H.M., 2004, Optimization of SRAP-PCR in Hot Pepper, Fenzi Zhiwu Yuzhong (Molecular Plant Breeding), 2(5): 689-693 (任羽, 王得元, 张银东, 李颖, 王恒明, 2004, 辣椒SRAP-PCR反应体系的建立与优化, 分子植物育种, 2(5): 689-693)
Wang X.Q., Zou Y.P., Zhang D.M., Zhang Z.X., and Hong D.Y., 1996, Problems in the use of RAPD to the study of genetic diversity and systematics, Zhiwu Xuebao (Acta Botanica Sinica), 38(12): 956-962 (汪小全, 邹喻苹, 张大明, 张志宪, 洪德元, 1996, RAPD应用于遗传多样性和系统学研究中的问题, 植物学报, 38(12): 956-962)
Wang Z.G., Zhang H.Y., Yu G.J., Zhang F., Wang Y.J., Xu Y., Guo S.G., and Gong G.Y., 2007, The orthogonal optimization of SRAP amplification system in Cucumber, Huabei Nongxuebao (Acta Agricultural Boreali-Sinica), 22(4): 112-115 (王振国, 张海英, 于广建, 张峰, 王永健, 许勇, 郭绍贵, 宫国义, 2007, 黄瓜SRAP反应体系的正交设计优化, 华北农学报, 22(4):112-115)
Wu W.H., Wang L., Cheng G.Z., Zhu Y.Y., and Pan Q.H., 2004, Studies on molecular genetics of rice blast fungus population——Comparison of genetic and pathotypic structures of two rice blast fungus populations derived from Guangdong and Yunnan provinces of China, Zhongguo Nongye Kexue (Scientia Agricultura Sinica), 37(5): 675-680 (吴伟怀, 王玲, 程贯忠, 朱有勇, 潘庆华, 2004, 稻瘟病菌群体的分子遗传学研究——广东省与云南省稻瘟病菌群体遗传及致病型结构的比较分析, 中国农业科学, 37(5): 675-680)
Wu Z.P., Yang W.X., Liu D.Q., and Zhang T., 2005, Establishment of SRAP technique system in wheat genome, Hebei Nongye Daxue Xuebao (Journal of Agricultural University of Hebei), 28(3): 66-69 (武志朴, 杨文香, 刘大群, 张汀, 2005, 小麦基因组SRAP扩增体系的初步研究, 河北农业大学学报, 28(3): 66-69)
Yang Q., and Zhang L.G. , 2007, Optimization of SRAP reaction system in Chinese Cabbage, Xibei Nongye Xuebao (Acta Agriculturae Boreali-occidentalis Sinica), 16(3): 119-123 (杨琦, 张鲁刚, 2007, 大白菜SRAP反应体系的建立与优化, 西北农业学报, 16(3): 119-123)






